資訊專欄INFORMATION COLUMN

此篇文章關鍵給大家介紹了Python完成一階矩馬爾可夫過程形成任意DNA序列實例詳細說明,感興趣的小伙伴可以參考借鑒一下,希望可以有一定的幫助,祝愿大家多多的發展,盡早漲薪。
1.基本原理
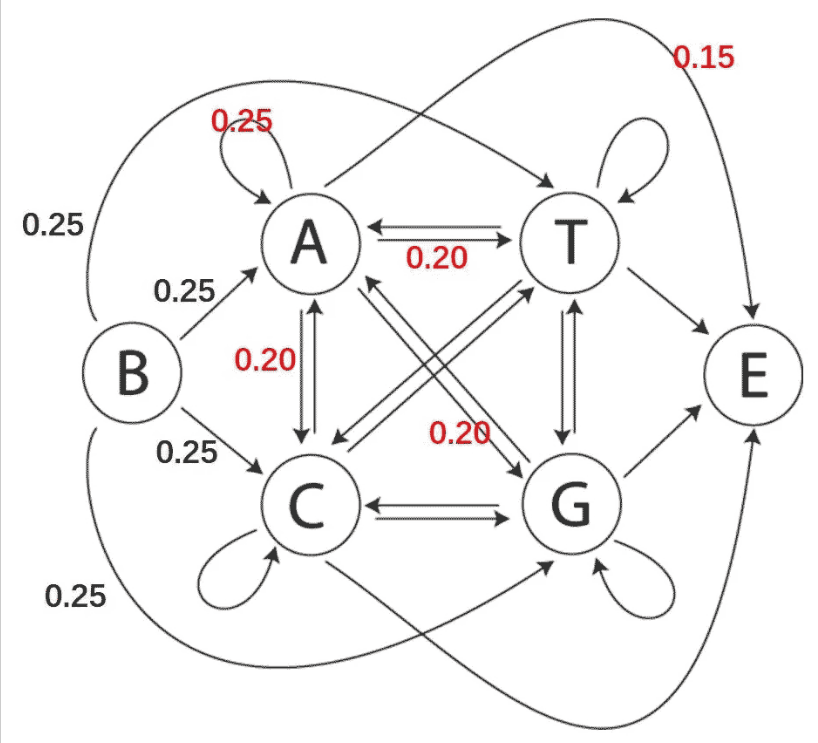
針對DNA序列,一階矩馬爾可夫過程可以看作現階段堿基對的種類僅在于上一位堿基對種類。如下圖1所示,1條編碼序列的開始(由B逐漸)有可能是A、T、G、C4種堿基對(且概率同樣,均是0.25),若編碼序列某還有一位A,則下一個堿基對是A、T、G、C的幾率依次為0.25、0.20、0.20、0.20,下一個無堿基對(即編碼序列完畢,情況為E)的幾率為0.15。
圖1 DNA序列的一階馬爾科夫鏈
2.代碼實現
以下代碼運行于Jupyter Notebook(Python 3.7);代碼功能是隨機生成一定數量的DNA序列,統計序列長度并繪制分布圖。若希望顯示隨機生成的序列,將代碼#print(''.join(Seq))前的#刪除即可。
import numpy
import random
import seaborn as sns
import matplotlib.pyplot as plt
#狀態空間
states=["A","G","C","T","E"]
#可能的事件序列
transitionName=[["AA","AG","AC","AT","AE"],
["GA","GG","GC","GT","GE"],
["CA","CG","CC","CT","CE"],
["TA","TG","TC","TT","TE"],]
#概率矩陣(轉移矩陣)
transitionMatrix=[[0.25,0.20,0.20,0.20,0.15],
[0.20,0.25,0.20,0.20,0.15],
[0.20,0.20,0.25,0.20,0.15],
[0.20,0.20,0.20,0.25,0.15]]
def RandomDNAs(Num):
max_len=0
i=0
Seq=[]#創建列表(Seq)用于添加堿基,以組成DNA序列
Len=[]#創建列表(Len)用于記錄每條生成序列的長度
while i!=Num:
Base=["A","G","C","T"]
START=random.choice(Base)#隨機從堿基中選擇一個作為序列的起始堿基
Seq.append(START)#將起始堿基添加至Seq中
while START!="E":
if START=="A":
change=numpy.random.choice(transitionName[0],p=transitionMatrix[0])
#以transitionMatrix矩陣第一行的概率分布隨機抽取transitionName第一行包含的事件
if change=="AA":
START="A"#如果轉移狀態是AA(即A堿基接下來的堿基是A,則將起始堿基設為A)
elif change=="AG":
START="G"
elif change=="AC":
START="C"
elif change=="AT":
START="T"
elif change=="AE":
START="E"
elif START=="G":
change=numpy.random.choice(transitionName[1],p=transitionMatrix[1])
if change=="GA":
START="A"
elif change=="GG":
START="G"
elif change=="GC":
START="C"
elif change=="GT":
START="T"
elif change=="GE":
START="E"
elif START=="C":
change=numpy.random.choice(transitionName[2],p=transitionMatrix[2])
if change=="CA":
START="A"
elif change=="CG":
START="G"
elif change=="CC":
START="C"
elif change=="CT":
START="T"
elif change=="CE":
START="E"
elif START=="T":
change=numpy.random.choice(transitionName[3],p=transitionMatrix[3])
if change=="TA":
START="A"
elif change=="TG":
START="G"
elif change=="TC":
START="C"
elif change=="TT":
START="T"
elif change=="TE":
START="E"
if START!="E":
Seq.append(START)#如果狀態轉移后不為End(E),則將轉移后的堿基加到Seq序列中
i+=1
Len.append(len(Seq))
if len(Seq)>max_len:
max_len=len(Seq)
#print(''.join(Seq))
Seq.clear()
plt.hist(numpy.array(Len),bins=max_len,edgecolor="white")
#顯示橫軸標簽
plt.xlabel("DNA Sequence Length")
#顯示縱軸標簽
plt.ylabel("Frequency")
#顯示圖標題
plt.title("Histogram of frequency distribution of DNA sequence length")
plt.show()
print("DNA序列的最大長度為:",max_len)
print("DNA序列長度的眾數為:",max(Len,key=Len.count))
%matplotlib notebook#若未使用Jupyter Notebook,此句不需要
RandomDNAs(1000)#1000表示隨機生成1000條序列3.運行結果
從以下4個序列長度分布統計可以看到,隨著隨機生成的序列數量增多,序列長度分布愈發集中,且長度為1bp的序列占比最多且逐漸增加。
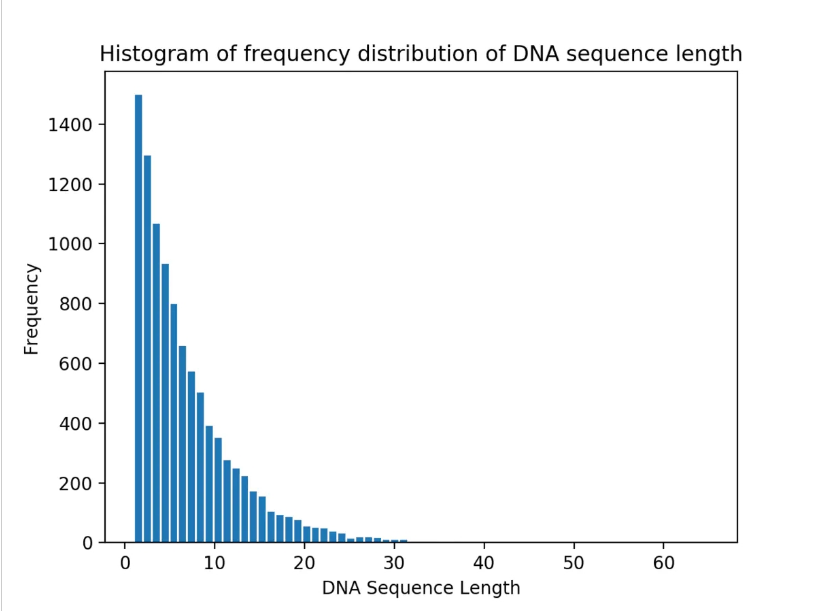
圖2 10,000條DNA序列的序列長度分布統計
10,000條DNA序列的序列中
DNA序列的最大長度為:65
DNA序列長度的眾數為:1
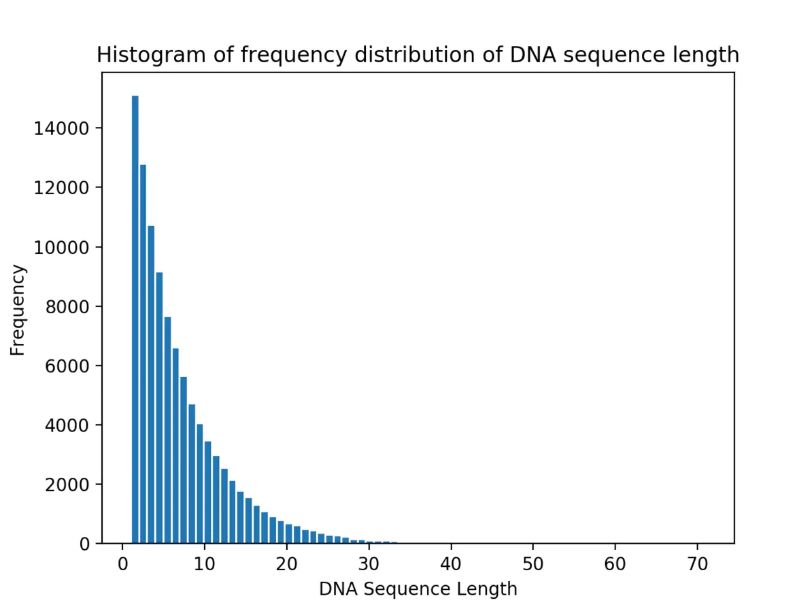
圖3 100,000條DNA序列的序列長度分布統計
100,000條DNA序列的序列中
DNA序列的最大長度為:71
DNA序列長度的眾數為:1
綜上所述,這篇文章就給大家介紹到這里了,希望可以給大家帶來幫助。
文章版權歸作者所有,未經允許請勿轉載,若此文章存在違規行為,您可以聯系管理員刪除。
轉載請注明本文地址:http://specialneedsforspecialkids.com/yun/128699.html
摘要:本文將討論兩種可用于解決貝葉斯推理問題的主要方法基于采樣的馬爾可夫鏈蒙特卡羅,簡稱方法和基于近似的變分推理,簡稱方法。而貝葉斯推理則是從貝葉斯的角度產生統計推斷的過程。貝葉斯推理問題還可能會產生一些其他的計算困難。 全文共6415字,預計學習時長20分鐘或更長 showImg(https://segmentfault.com/img/bVbvFZZ?w=1280&h=853); 圖片來...
摘要:近來在深度學習中,卷積神經網絡和循環神經網絡等深度模型在各種復雜的任務中表現十分優秀。機器學習中最常用的正則化方法是對權重施加范數約束。 近來在深度學習中,卷積神經網絡和循環神經網絡等深度模型在各種復雜的任務中表現十分優秀。例如卷積神經網絡(CNN)這種由生物啟發而誕生的網絡,它基于數學的卷積運算而能檢測大量的圖像特征,因此可用于解決多種圖像視覺應用、目標分類和語音識別等問題。但是,深層網絡...
摘要:通過將神經元的值設置為希望的模式來訓練網絡,之后可以計算權重。輸入神經元在完整網絡更新結束時變成輸出神經元。在某種程度上,這類似于峰值神經網絡,并不是所有的神經元始終都在發射并且點的生物合理性得分。 隨著新的神經網絡架構不時出現,很難跟蹤這些架構。知道所有縮寫(DCIGN,BiLSTM,DCGAN,任何人?)起初可能有點壓倒性。 所以我決定編寫一個包含許多這些體系結構的備忘單。這些大多...
閱讀 911·2023-01-14 11:38
閱讀 878·2023-01-14 11:04
閱讀 740·2023-01-14 10:48
閱讀 1982·2023-01-14 10:34
閱讀 942·2023-01-14 10:24
閱讀 819·2023-01-14 10:18
閱讀 499·2023-01-14 10:09
閱讀 572·2023-01-14 10:02